오늘은 R 코드를 병렬 처리하는 방법에 대해서 알아보겠습니다. 병렬 처리? 많이 들어보셨던 말씀이겠지만 병렬 처리가 일단 무엇인지 먼저 알아보고 진행해보도록 하겠습니다.
Parallel Processing
병렬 처리 혹은 계산은 컴퓨터를 이용해서 동시에 많은 계산을 하도록 하는 처리 방법 중에 하나입니다. 과거에는 프로세서 성능이 하나당 코어 한 개를 가지고 있기 때문에 병렬 처리라는 것은 사실상 불가능한 것에 가까웠습니다. 하지만 이를 가능하도록 한 것은 멀티태스킹 방식이 있었기 때문이죠. 이 방법은 하나의 계산을 여러 개로 쪼개서 나눠 처리하는 방식으로 동시에 많은 계산을 한꺼번에 처리하는 것이랑은 약간의 차이가 있었습니다.
하지만 현대 프로세서에는 한 프로세서에 여러 개의 코어가 달려 있는 프로세서가 많이 나오고 있죠. 대표적으로 8개의 코어를 달린 녀석을 옥타 코어 프로세서라고도 많이 이야기를 합니다. 우리는 이러한 많은 코어를 가지고 수많은 반복 연산을 각 코어만큼 나눠 분산하는 처리 방식을 바로 병렬 처리라고 이야기 합니다.
사실 여기서 말하는 옥타 코어는 우리가 흔히 쓰는 노트북, 데스크톱 중에서 가장 성능이 높은 프로세서를 이야기 합니다. 여러분들이 사용하는 PC에 따라서 프로세서가 듀얼 코어일 수도 있고, 쿼드 코어일 수가 있습니다. 하지만 정말 오래된 컴퓨터가 아니라면 싱글 코어를 가지는 경우는 거의 없죠… 그러니 얼마든지 병렬 처리 실습을 하는 데 있어 큰 문제가 되지 않을 것이라고 판단합니다.
When should use?
그렇다면 우리는 이러한 병렬 처리를 어떨 때 사용해야 할까요? 대표적으로 저는 병렬 처리를 서버 애플리케이션과 같은 여러 명의 사용자가 반복적인 작업을 요청할 때 많이 사용했었습니다. 하지만 R을 사용해서 데이터 처리를 할 경우에는 수많은 데이터 집합체를 반복해서 처리할 때 많이 사용하죠. 데이터가 커지거나 그 양이 많아지면 반복하는 횟수도 많아지니깐요.
1 | len <- list.files("~/images", pattern = "\\.png$") |
우리가 흔히 이미지 처리를 하려고 자신의 스토리지에 있는 이미지를 꺼내 모든 이미지를 Grayscale 처리를 하려고 한다면 보통은 이러한 방법을 사용할 것입니다. (위의 코드는 설명을 쉽게하기 위해 반복문을 사용한 것입니다.)
1 | lapply(len, function(x) { |
R을 좀 사용해보신 분들이라면 이러한 방법을 사용해도 되겠지요? 반환 값은 해당 파일이 어떻게 처리되었는지에 대한 마지막 메시지를 리스트로 반환해주기 때문에 처리도 매우 유용할 것이라고 생각합니다. 하지만 여전히 똑같은 것은 같은 동작을 계속 반복한다는 것입니다.
예를 들어 저기에 있는 이미지가 100,000개 있다고 생각하여 보겠습니다. 평상 반복문은 맨 첫 이미지가 반복문의 코드를 처리하고 끝날 때까지 다음 이미지는 계속 대기 중인 상태로 머물게 됩니다. 하지만 우리는 코어를 여러 개 가지고 있기 때문에 쉬고 있는 코어를 사용한다면 더욱 일 처리가 빨라지겠죠?
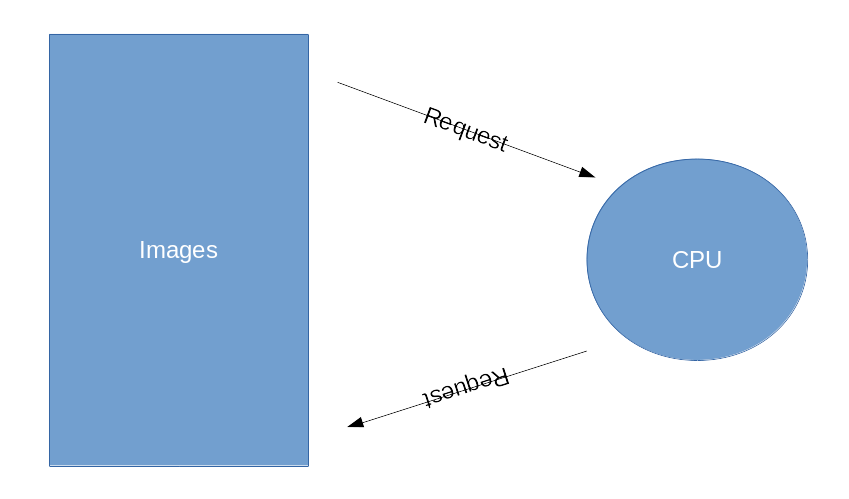
위 이미지는 한 개의 프로세서를 사용했을 때 반복문을 처리하는 모습입니다. Images가 처리해야 하는 데이터라고 한다면 프로세서가 한 개일 떄, 하나의 작업을 요청하고 그 작업이 끝나면 다음 파일을 CPU가 요청하는 방식인 것이죠.

프로세서가 여러 개인 경우에는 이렇게 놀고 있는 코어의 녀석들이 일거리를 달라고 외치게 됩니다. “나 놀고 있으니, 일 줘”라는 것이죠. 이렇게 하면 혼자서 처리하는 것보다 일의 효율이 더 좋아지니 수없이 많은 데이터에 대해서도 빠르게 처리할 수 있는 효과가 생기는 것입니다.
How to use
그렇다면 우리는 어떻게 이 코어들을 사용할 수 있을까요? R에서 여러 개의 코어 혹은 프로세서를 이용하여 병렬 처리를 도와주는 parallel 패키지가 존재합니다.
1 | install.packages('parallel') |
하지만 이 패키지를 사용해서 우리가 사용하는 일반적인 R의 반복문을 가지고 병렬 처리할 수 있는 것은 아닙니다. 따라서 우리는 병렬 처리용 반복문 패키지를 한 가지 더 설치해야 합니다.
그럼 이제 본론으로 넘어가보도록 하죠.
1 | cores <- parallel::detectCores() - 1 |
R에서 병렬 처리를 할 때는 먼저 자신이 몇 개의 코어를 사용해줄 것인지 정하고 해당 코어의 갯수만큼 클러스터화를 해줘야 합니다.
Cluster
클러스터는 여러 개의 분산된 시스템이나 코어를 마치 하나의 시스템 혹은 코어처럼 사용해주는 것을 일컫는 단어입니다.
Parallel 패키지에서는 Cluster를 두 가지 방식으로 구성할 수 있습니다.
- FORK 방식 (not support CP/M)
- SOCK/PSOCK 방식
detectCores의 함수는 자신이 현재 사용하고 있는 PC의 코어 갯수가 몇 개인지를 반환하는 함수입니다. 보통 Linux / OS X에서는 /proc 라는 커널 정보에서 가져오게 되고 Windows에서도 커널 객체 함수에서 CPU 코어의 갯수를 가져오는 함수가 제공되기 때문에 이를 이용할 것입니다.
1 | parallel::parLapply(cl = cluster, x = len, function(x) { |
앞서 말씀드렸 듯이 병렬 처리를 할 때는 for문처럼 일반 구문을 사용하시면 안됩니다. 이는 apply 함수 계열도 동일하게 적용됩니다. 따라서 lapply 함수를 병렬 처리할 때는 parallel 패키지에서 제공하는 parLapply 함수를 사용하셔야 합니다.
그 이유는 병렬 처리 전용 함수에서 사용할 클러스터 시스템 하나를 정해줘야하기 때문입니다. 그렇지 않으면 일반적인 코어 한 개를 사용하게 되기 때문이죠.
그런데 한 가지 주의 사항이 있습니다. 병렬 처리된 코드는 전역 변수라 하더라도 등록되지 않은 변수에 대해서는 해당 메모리에 접근할 수 없습니다. 코드로 예시를 들어보도록 하죠..
1 | ex <- 5 |
위의 코드를 보면 병렬처리된 코드 파라미터로 images 파일이 들어가있습니다. 거기에 추가로 내가 함수 밖에서 선언한 ex를 사용하려 하면? 오류가 발생하게 됩니다. 왜 그럴까요?
병렬 처리된 코드는 해당 클러스터 프로세스 안에서 동작하게 됩니다. 즉 현재 자신이 사용하고 있는 시스템과 다른 시스템을 소켓이나 Fork를 통해서 다른 프로세스로 만들어준 후 진행하기 때문에 이럴 경우 현재 사용하고 있는 메모리의 주소를 클러스터 프로세스에 던져줘야 합니다.
1 | parallel::clusterExport(cluster, "ex") |
이렇게 자신이 사용할 외부 메모리 주소를 클러스터 쪽으로 던지면 해당 메모리 주소에 있는 값이 복사되어 클러스터 쪽에 서도 변수값을 사용할 수 있게 됩니다.
그런데, 이번엔 library 함수로 불러온 패키지를 사용해야 하는데, 이 역시 되지 않네요. 클러스터 공간에 이 패키지를 사용할 수 있도록 해줘야합니다.
1 | parallel::clusterEvalQ(cluster, library(dplyr)) |
그럴 때는 clusterEvalQ 함수를 사용해서 원하는 클러스터에 library 함수를 불러오시면 됩니다.
1 | parallel::stopCluster(cluster) |
병렬 처리가 마치고 난 다음에는 자신이 사용한 코어를 반드시 운영체제에 반납해줍니다. 그렇지 않으면 해당 Rsession이 계속 리소스를 잡고 있게 됩니다.
Using foreach
apply 계열 함수에 대해서는 parallel 패키지에서 제공하는 함수로 충분히 병렬 처리가 가능했습니다. 그렇다면 for문을 사용하여 병렬 처리하는 방법은 없는 건가요?
1 | install.packages(c('foreach', 'doParallel')) |
for문을 사용한 병렬 처리도 당연히 가능합니다. 우리는 이를 위해서 foreach와 doParallel이라는 두 가지 패키지를 추가로 설치해야 합니다.
foreach
foreach 패키지는 Loop와 lapply 함수를 섞어 만든 패키지입니다. loop 문법을 사용하면서 리스트, 데이터 프레임, 벡터 등 원하는 자료형으로 반환할 수 있다는 특징을 가지고 있어 R에서 매우 인기가 많은 패키지로 유명합니다.
1 | # 사용할 코어 갯수와 클러스터 선정 |
foreach문은 다양성이 많이 존재하는 패키지입니다. 왜냐하면 개발자 자신이 원하는 반환값을 고르거나 반복문안에서 사용할 패키지, 외부 변수를 정해줘야할 수가 있기 때문이죠. foreach 패키지는 parallel 패키지와 다르게 한 코드에서 모든 것을 처리한다는 것이 특징입니다.
1 | # 이미지 전처리 함수 |
만약 예를 들어 병렬 처리하는 코드 영역에 dplyr 패키지의 코드를 적고싶다면 library 함수를 사용해서 include 시키는 것이 아닌 함수 파라미터에 dplyr를 적어주시면 됩니다. 해당 파라미터는 벡터 형태로 동작하기 때문에 여러 개의 패키지를 정할 경우에는 벡터 형식을 사용하면 됩니다. 또한 내가 따로 정한 함수를 사용할 때도 마찬가지입니다. 해당 함수를 클러스터 공간에 던져줘야 하는데, 이 때는 .export 파라미터를 사용합니다. 이 파라미터 역시 벡터 형식을 사용합니다.
병렬 처리하는 동안 내가 처리하고 있는 프로세스가 어디까지 진행되는지 알 수 있는 방법은 없을까?
우리는 반복문을 사용하고 이 처리가 어디까지 진행되었는지 확인하기 위해 ProgressBar를 사용했을 것입니다.
1 | tpb <- txtProgressBar(min = 0, max = len, style = 3) |
평상적으로 반복문을 사용했을 때 ProgressBar를 사용하면 이러한 형태로 사용했을 것입니다. 하지만 foreach의 병렬 처리를 사용할 때 이 코드는 동작하지 않습니다.
우리는 이 코드를 동작시키기 위해 doSNOW 패키지를 사용해야 합니다.
1 | install.packages('doSNOW') |
doSNOW 패키지는 병렬 처리하는 클러스터 내에서 백엔드 역할을 해주는 패키지입니다.
1 | # 원하는 클러스터 선정 |
백엔드에서 처리할 함수를 생성하고 이를 리스트에 넣은 다음 클러스터에 적용하면 손쉽게 병렬 처리하는 클러스터 내에서도 병렬 처리의 진행 상황을 시각적으로 확인할 수 있습니다.
Debugging
R에서 특정 시점을 디버깅할 때는 아래의 방법을 사용합니다.
1 | debug(preProcessing) |
하지만 병렬 처리를 이용할 때는 어떠한 방법으로 디버깅을 할 수 있을까요? 함수 안에 병렬 처리가 있을 경우 debug 함수로 디버깅 옵션을 부여했다 하더라도 해당 클러스터 구간은 디버깅되지 않고 처리될 때까지 프로세서가 대기하는 현상이 발생하여 디버깅 작업이 동작하지 않습니다.
그럴 때는 두 가지 방법이 있습니다.
- try-catch 문을 사용하여 반환 데이터에 오류 메시지를 출력하는 방법
- output file을 생성하는 방법
tryCatch 문을 사용하는 방법은 apply 계열 함수 및 foreach 문에서 동일하게 적용할 수 있는 방법입니다. 하지만 output file을 통해 로그를 출력하는 방법에는 각각 사용법에 차이가 있습니다.
try-catch 문의 사용
먼저 foreach 문을 보도록 하겠습니다.
1 | foreach::foreach(i = 1:len) %dopar% { |
기본적으로 foreach 패키지는 리스트를 변환하기 때문에 만약 오류가 발생한 경우라면, 해당 번째의 리스트에 에러 메시지를 남기므로 이를 통해서 디버깅할 수 있습니다.
1 | parallel::parLapply(cl = cluster, x = len, function(x) { |
lapply계열에서도 동일하게 적용하면 됩니다.
output file 생성
output file을 생성하는 것은 Cluster를 생성할 때 지정할 수 있습니다.
1 | cluster <- parallel::makePSOCKcluster(names = cores, outfile = "~/clusterlog.txt") |
클러스터 생성 함수에 outfile 옵션을 주고 출력할 파일 경로를 지정해주면 처리되는 로그가 파일에 출력됩니다.
만약 각 이미지별로 처리되는 로그를 각각 보고싶다면..
1 | ... |
cat 함수로 메시지를 출력하되, 이를 파일에 쓰도록 유도하면 됩니다.
Caching
병렬 처리에서 캐싱은 대용량 처리시 매우 중요합니다. 여러분들이 병렬 처리를 하러 검색을 하시게 된다면 그것은 분명히 양이 많기도 하겠지만 처리할 데이터의 크기가 매우 큼을 이야기할 것입니다.
캐싱은 왜 사용하는 것일까요? 서버 애플리케이션을 개발했을 때 토큰과 같은 자주 사용하는 데이터들은 주로 캐싱을 사용하여 빠르게 불러오곤 했었습니다.
병렬 처리에서도 캐싱을 이용한다면 더 빠른 처리를 만나보실 수 있을 것입니다.
1 | install.packages('digest') |
이를 위해 우리는 digest 패키지를 이용해야 합니다. digest 패키지는 R의 객체를 압축하여 해싱하여 RData 파일로 저장하도록 하는 패키지입니다.
1 | # 클러스터 생성 |
병렬 처리를 함에 있어서 중복된 처리는 당연히 발생할 수 있습니다. 예를 들자면 100,000개의 이미지 중에서 일부 이미지처리 중에 오류가 발생했다면 이를 다시 실행해야 하는데, 그럴 경우, 이미 처리된 이미지까지 다시 처리해야 하는 문제가 생기게 되고 그만큼의 시간이 소모됩니다. 하지만 캐싱된 파일을 불러온다면? 그 이미지 처리하는 만큼 파일을 읽는 시간으로 단축시킬 수가 있게 되는 것이죠.
Work Load
Computer Science에서는 한 작업이 작업을 위해 일정량의 리소스를 운영체제로부터 할당받게 됩니다. 그런데, 병렬 처리를 할 때는 작업 이벤트가 동시에 발생하기 때문에 서로가 자원 확보를 위해 나서게 될텐데 이들을 경쟁시켜서는 안됩니다. 그렇게 될 경우 누구는 적게 받고, 누구는 많이 받기 때문에 서로가 정해진 자원을 균등하게 배분시키는 것이야 말로 병렬 처리의 핵심입니다.
parLapply와 foreach 함수는 모두 Wrapper function이고 실제로는 병렬 처리에 아무런 관련을 하지 않기 때문에 이들을 수정한다고 해서 되지는 않습니다.
Wrapper Function
랩핑 함수를 가리키는 이 단어는 실질적으로 작업을 하는 함수가 해당 작업의 주원천이 아니고 다른 함수의 기능을 빌려 일을 처리하는 함수를 말합니다.
1 | parLapply <- function(cl = NULL, X, fun, ...) { |
위 코드는 parLapply 함수의 원천 코드입니다. 자세히 보면 우리가 주어주는 데이터인 X를 splitList를 통해 리스트로 분할하여 이를 Cluster가 가지고 있는 노드만큼 자원을 균등하게 분배합니다.
그런데, 이렇게 작업을 균등하게 분배하면 워크 로드의 문제는 발생하지 않을 것 같은데요?
만약 모든 데이터가 똑같은 함수로 처리한다고 하더라도, 위에서 설명한 캐싱을 이용한다면 워크 로드의 문제는 발생할 수 밖에 없습니다. 왜 그럴까요?
바로 캐싱된 데이터에 대해서는 그 작업량이 많이 줄어들기 때문입니다.
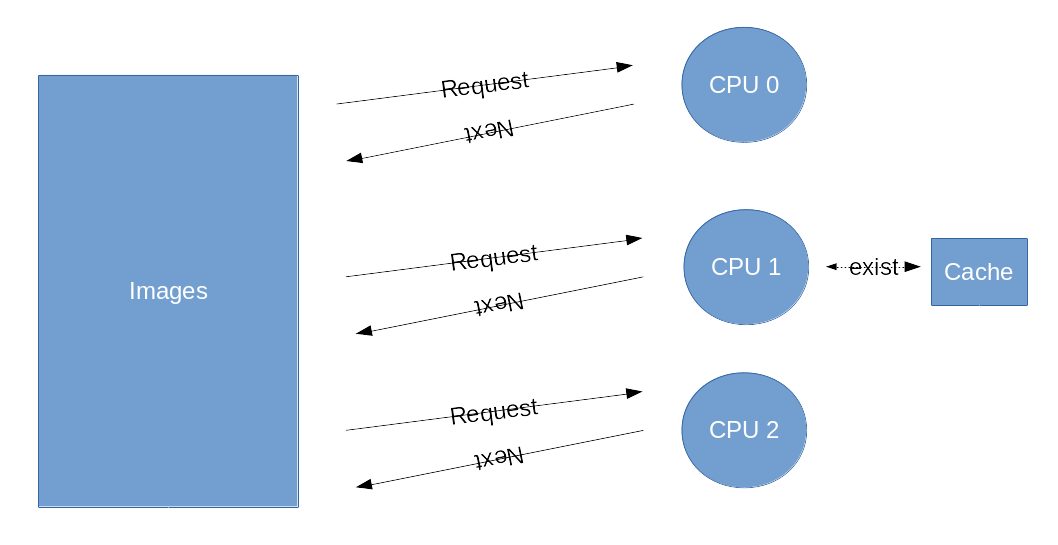
위 이미지를 한 번 보면, 3개의 코어 중 2개은 캐시가 없어 계산을 수행하고 있지만 1개는 cache에 이미 처리한 데이터가 존재하기 때문에 있는 것을 가져다주므로 작업량이 훨씬 줄어들게 됩니다.
Memory Load
저는 R을 이용해서 이미지 처리를 자주 하고 있습니다. 그럴 때마다 항상 매번 민감하게 다루는 것이 바로 메모리 부하 문제입니다. 물론 좋은 서버가 있어 해당 서버로 돌리면 그렇게 큰 문제가 생기진 않지만 개발자로써 메모리를 가능한한 가장 최적화 시키는 것은 누구나 가지고 싶어하는 역량 중 하나일 것입니다.
유독 병렬 처리할 때는 이 메모리에 신경을 많이 써야합니다. 프로세서는 내가 현재 가지고 있는 코어에서 1개 정도만 사용하지 않아도 시스템 유지에 큰 문제가 없지만 메모리는 그러한 limit을 커널에서 정하는 거 빼고는 없기 때문입니다. 커널에서 정하면 적어도 시스템 크래쉬는 방지할 수 있어도 내가 돌리는 코드가 원활히 돌 것이라는 보장은 할 수 없기 때문입니다.
그래서 OS X / Linux에서는 ForkCluster를 사용할 수 있습니다. ForkCluster는 SOCK에 비해 그 메모리 소모량이 많이 적습니다. 메모리가 한계치에 이를 경우 Fork의 경우 쓸모없는 변수를 버릴 때 별도의 시간이 핋요하지도 않고 그에 따른 메모리의 소모가 발생하지 않습니다. 그래서 안전하게 변수의 메모리를 해제할 수 있는 아주 좋은 조건입니다.
또한 R에서는 별도의 코드 없이는 사용했던 변수를 메모리에서 해제하지 않습니다. 그 이유는 바로 스크립트 언어이기 때문입니다. C나 Java와 같은 코드는 컴파일 이후 프로그램이 종료되면 모든 변수의 데이터를 버리고 사용한 메모리를 반납하지만 R은 session이 종료될 때까지 절대로 사용한 메모리를 반납하지 않기 때문에, 반드시 아래와 같은 방법으로 사용한 메모리를 반납하는 것이 매우 중요합니다.
1. rm 함수의 사용.
1 | rm(img) |
2. Garbage Collection 호출
1 | gc() |
gc 함수는 JVM을 사용하셨다면 많이 보셨을 함수일 것입니다. 대부분 R에서도 자동적으로 이 함수를 많이 부르긴 하지만 이 함수의 알고리즘이 그렇게 좋은 편은 아닙니다. 따라서 병렬 처리시에는 수동으로 호출하는 것도 나쁘지 않은 방법입니다.
3. 남은 메모리에 따라 코어 수 분배
parLapply 함수를 보셨다시피 모든 작업은 균등하게 분배됩니다. 이 말은 균등하게 메모리 양도 적절히 분배된다는 이야기이기 때문에 만약 이 작업을 모두 수행하는 데 충분한 메모리 용량이 아니라고 판단된다면 메모리 용량에 따라 코어 수를 분배하는 것도 나쁘지 않은 방법입니다.
1 | cores <- memory.limit() / memory.size() |
Linux에서 이러한 방법을 사용할 때는 주의 사항이 있습니다. 보통 커널 튜닝을 진행하지 않고 이러한 방법을 사용할 경우 memory.limit 함수를 불러올 때 Inf라는 결과값을 출력하게 되는데, 이 때는 현재 자신의 시스템의 메모리 제한이 걸려 있지 않다는 것입니다. 이러한 경우에는 아래의 명령어를 이용해서 프로세스당 메모리 제한을 반드시 진행해줘야 합니다.
1 | ulimit -t 600 -v 4000000 |
마치며…
R을 사용하여 병렬 처리하는 포스트를 끄적여 봤습니다. 사실 큰 데이터의 처리를 다룰 일이 많이 없다고 생각했는데, 막상 다뤄보니 Python 못지 않게 R이 좀 더 대용량 처리에 더 견고하게 다가갈 수 있음을 알 수 있었습니다. 물론 Python이 데이터 처리에 그렇게 큰 문제가 있다는 것은 아닙니다. 서로가 장단점을 가지고 있었는데, 간단히 요약해보자면 아래와 같습니다.
Python
- 대용량 처리를 위해 Pandas, Numpy 등이 제공되어 있다.
- 원래 프로그래밍 언어여서 이러한 데이터 처리와 함께 애플리케이션을 쉽게 개발할 수 있다.
하지만 numpy, pandas가 끝이고 아직 pandas가 대용량을 잘 처리해줄 만큼의 큰 요소는 가지고 있지 않다. - pyspark라는 걸 이용해서 데이터 처리한다면 좀 더 유용하게 사용할 수 있을지도 모르겠다.
R
- 톹계, 데이터 처리 및 분석이 용이한 언어이다.
- C++ 라이브러리, Java 라이브러리 등 Python과 똑같이 여러 언어를 공용해서 사용할 수 있다.
- 원래 목적이 프로그래밍 언어가 아니다보니 애플리케이션을 개발하는 데는 한계점이 있다.
하지만 최근 Shiny라는 R의 웹 프레임워크가 급속으로 발전하고 있긴하다. - 무엇보다 속도가 느려서 지속적인 서비스 애플리케이션을 개발하는 데는 부족함이 많다.
사실 이전까지만해도 데이터 처리를 Python으로 해보겠다는 생각이 강했습니다. 하지만 R을 사용해서 이것저것 데이터 처리를 해보면서 데이터 전처리나 통계 등을 다른 언어보다 쉽게 처리할 수 있는 언어는 R 뿐이라고 생각합니다. 그리고 무엇보다 Pandas에 비해 Data frame 사용이 쉽고 인코딩에 강력해서 다른 DBMS와의 연동도 오히려 더 쉽다는 생각이 들었습니다.